



免疫组库测序打开药物研发新视野(一):什么是免疫组库测序?、免疫组库测序打开药物研发新视野(二):免疫组库检测的方法有哪些?两篇文章介绍了免疫组库的基本概念、人体内免疫组库多样性产生的生物学过程及免疫组库常用检测方法。本文将重点介绍免疫组库测序在TCR相关的肿瘤免疫治疗中的应用,并对检测结果进行生物学意义解读。
PART 01
TCR在肿瘤免疫治疗中发挥核心作用
TCR(T细胞受体)是适应性免疫系统中的关键受体,它们在肿瘤治疗中发挥核心作用。TCR主要存在于T细胞表面,负责识别并结合由抗原呈递细胞(如巨噬细胞、树突状细胞等)通过MHC分子(主要组织相容性复合体)呈递的多肽片段。当TCR与抗原-MHC复合物结合后,会触发T细胞活化和增殖,从而分化为不同的效应子T细胞亚群,包括CD8+ 细胞毒性T细胞和CD4+ 辅助T细胞。
CD8+ 细胞毒性T细胞(CTLs)能够特异性识别并消灭表达异常抗原的肿瘤细胞。它们通过与肿瘤细胞表面的主要组织相容性复合体(MHC)Ⅰ类分子结合而被激活,该复合体呈递的是来源于肿瘤细胞内部的多肽片段。当CD8+ T细胞识别到这些抗原后,它们会释放穿孔素、颗粒酶和其他效应因子,导致靶向的肿瘤细胞裂解死亡。
CD4+ 辅助T细胞(Th cells)能够识别抗原呈递细胞(如树突状细胞)上MHCⅡ类分子结合的肽段,并通过与这些细胞相互作用而被激活。激活后的CD4+ T细胞可以分化为不同的辅助亚型,包括Th1、Th2、Th17等,分别分泌不同类型的细胞因子来激活和调控其他免疫细胞。并且通过分泌细胞因子来调节其他免疫细胞的功能,包括促进B细胞产生抗体(参与体液免疫),以及激活巨噬细胞、自然杀伤细胞等进行细胞介导的免疫反应,这对于疫苗诱导的免疫应答至关重要。
肿瘤免疫治疗通过增强患者的免疫系统识别和攻击肿瘤细胞的能力,或克服肿瘤细胞免疫逃逸机制,从而达到控制、缩小甚至消除肿瘤的目的,包括但不限于免疫检查点抑制剂、CAR-T细胞疗法、肿瘤疫苗以及肿瘤浸润淋巴细胞(TILs)疗法等。肿瘤免疫治疗通过多种机制直接影响免疫系统中各类免疫细胞的数量、种类和功能状态,TCR免疫组库测序可以检测肿瘤特异性的T细胞克隆、评估肿瘤微环境的免疫状态、评估免疫治疗效果、监测治疗前后的免疫应答变化,并且可以辅助预测治疗反应与患者预后。
PART 02
单个样本TCR免疫组库测序结果
熙宁|精翰NGS实验室开发了成熟的免疫组库测序生信分析流程,对检测样本进行 T 细胞受体基因克隆鉴定检测,并对受检者的不同时间节点的样本进行 T 细胞受体基因克隆鉴定检测,对特定的TCR 的克隆比例,克隆多样性、V/J 基因的使用频率进行统计,识别样本间的共有克隆,并追踪不同样本 TCR 指标的动态变化,如克隆总数、克隆类型、多样性指数、克隆丰度等,辅助评估免疫治疗的临床效果。
本文将以TRB为例,从克隆多样性、CDR3长度分布、V-J基因使用频率分布等方面对单个样本的TCR免疫组库测序结果进行展示,并从生物学角度对检测结果进行解读(注:图示中所有数据均来自虚拟样本)。

2.1 克隆多样性
计算样本TCR组成多样性的指标有Shannon’s entropy,Inverse Simpson’s index,Evenness,Clonality。
• Shannon’s entropy 计算公式:
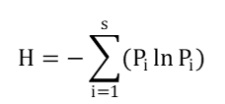
• Inverse Simpson’s index 计算公式:
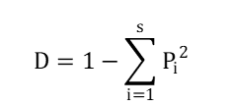
• Evenness 计算公式:
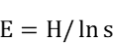
• Clonality 计算公式:
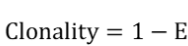
其中:s 表示实际观测到的 TCR 重组序列的数目;Pi表示第 i 个 TCR 重排序列在所有 TCR 中所占的比例。
Shannon 和 Inverse Simpson 值越大,说明样本的 TCR 多样性越高。Evenness 的取值范围为 0~1,指样本的均匀度。 Clonality 的取值范围为 0~1,指样本的克隆性。

TCR克隆多样性与年龄呈显著正相关,随着年龄的增加TCR的多样性呈现下降趋势。TCR多样性的异常增高,与免疫系统训练不充分有关;TCR多样性的异常减少与免疫缺陷、免疫系统发育不全以及肿瘤性或反应性T细胞大量扩增有关。

2.2 CDR3 长度分布
在某些疾病状态下,例如自身免疫性疾病、某些感染疾病(如HIV感染)以及癌症,可能会观察到TCR CDR3长度谱的改变。比如,在某些肿瘤患者中,可能会发现TCR CDR3的长度分布出现偏移,这可能是由于肿瘤特异性T细胞克隆扩增或者免疫反应失衡所导致。此外,某些病毒感染后,为了应对病毒抗原变异,T细胞库可能会产生更多具有特定CDR3长度的克隆。
统计样本内CDR3区域的核苷酸序列的长度分布与频率的关系,柱状图分别由频率最高的20种CDR3氨基酸序列与V片段填充,结果分别见图1与图2:
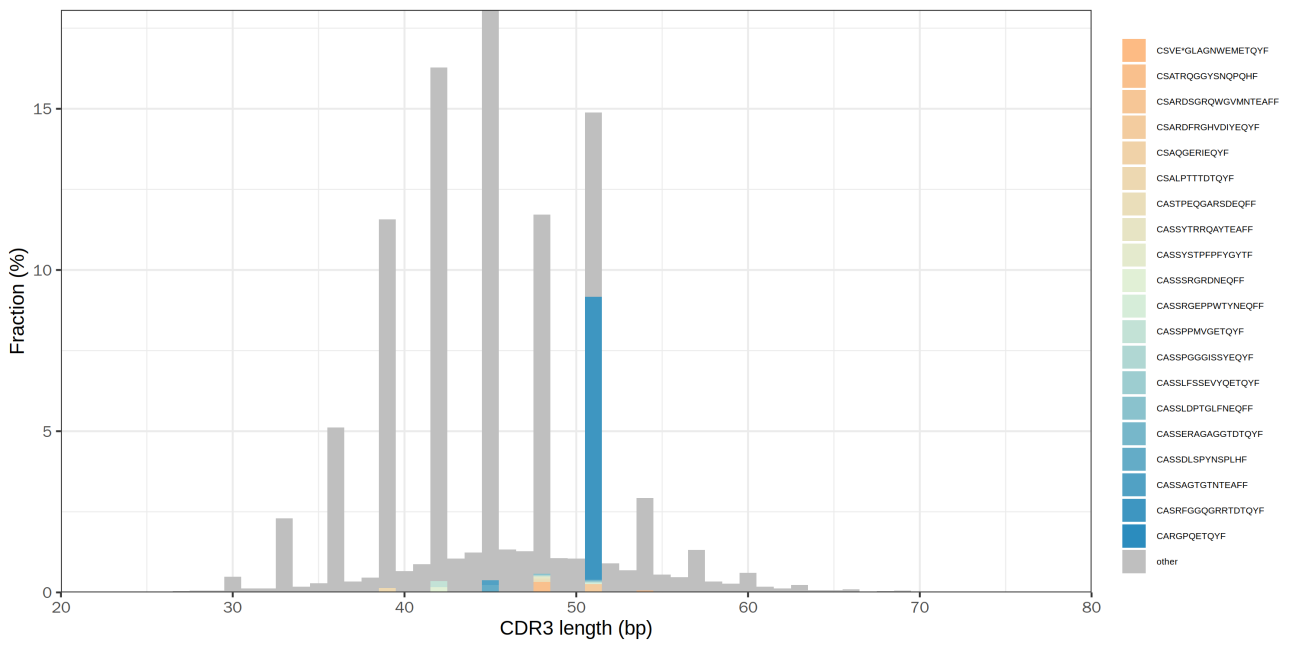
图1 CDR3 核苷酸序列长度与Top20 序列分布直方图
上图中横坐标表示CDR3核苷酸序列的长度,纵坐标表示对应长度CDR3数量在所有CDR3的比例,展示频率最高的20种CDR3的多肽序列。
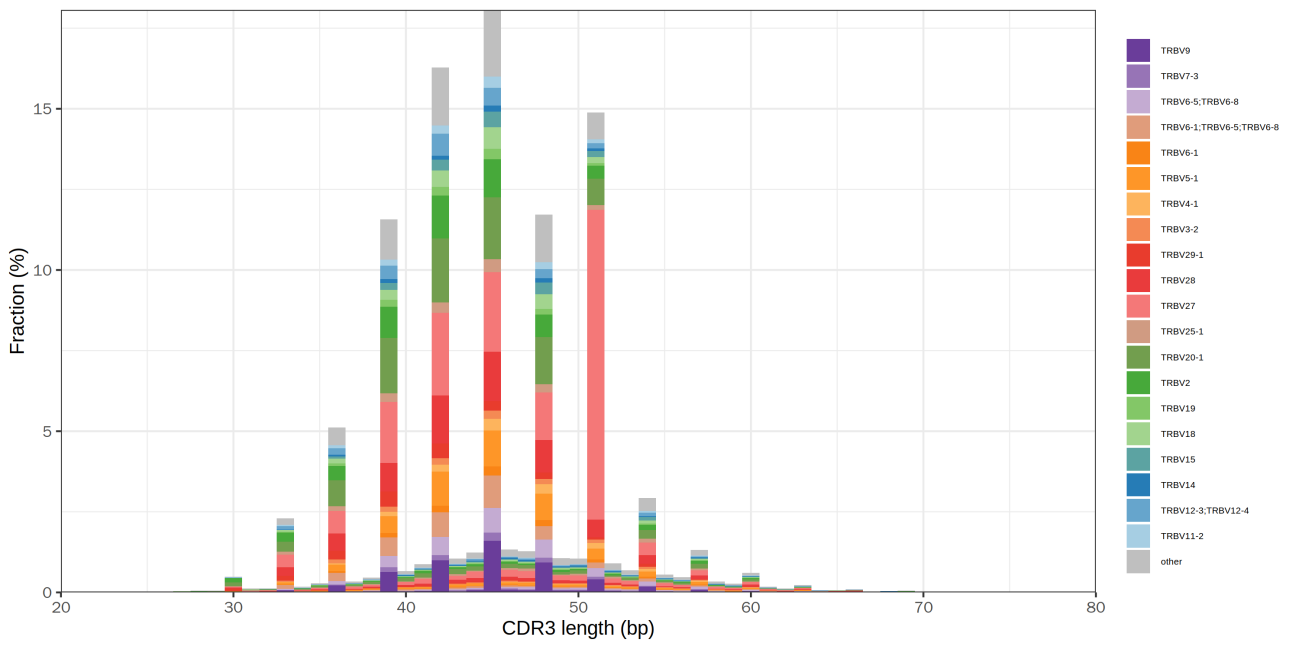
图2 CDR3 核苷酸序列长度与Top V片段分布直方图
上图中横坐标表示CDR3核苷酸序列的长度,纵坐标表示对应长度CDR3数量在所有CDR3的比例,展示频率最高的 20 种 V 片段的分布。

2.3 V/J片段使用频率统计
截止到2024年2月26日,根据IMGT数据库的统计,人类基因组总共包含 237-243 个 TR 基因,具体基因数取决于单倍体类型。每个单倍体基因组的功能性 TR 基因数量为 172-185。TR基因在染色体上的分布及各片段的数目统计如下表:

统计样本内V片段与J片段的使用频率,结果分别见图3与图4,并统计V-J对的组合与使用频率,结果见图5与图6:
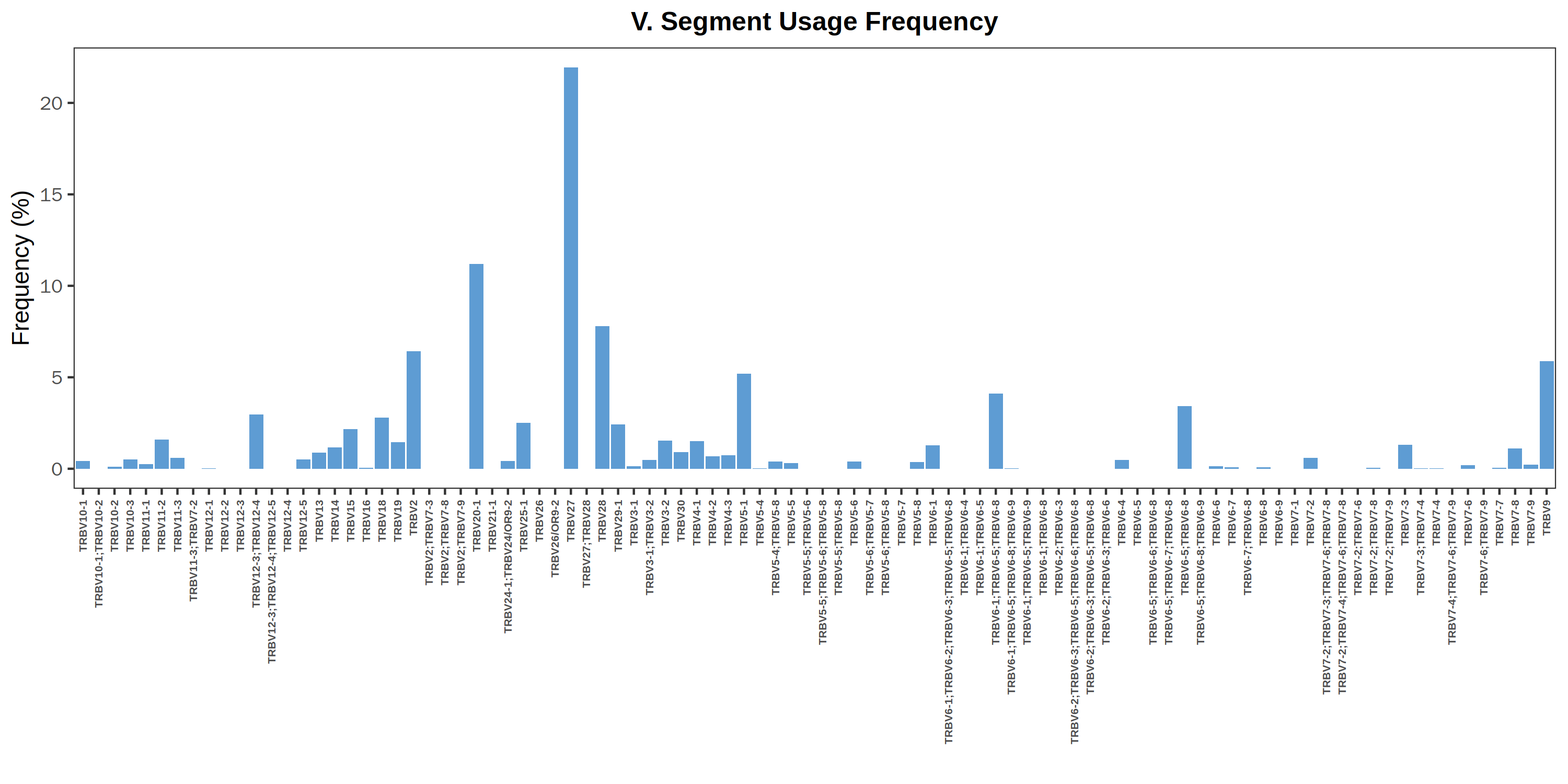
图3 V 基因使用频率分布直方图
上图中横坐标表示不同的V片段的名称,纵坐标表示对应V片段的使用频率。
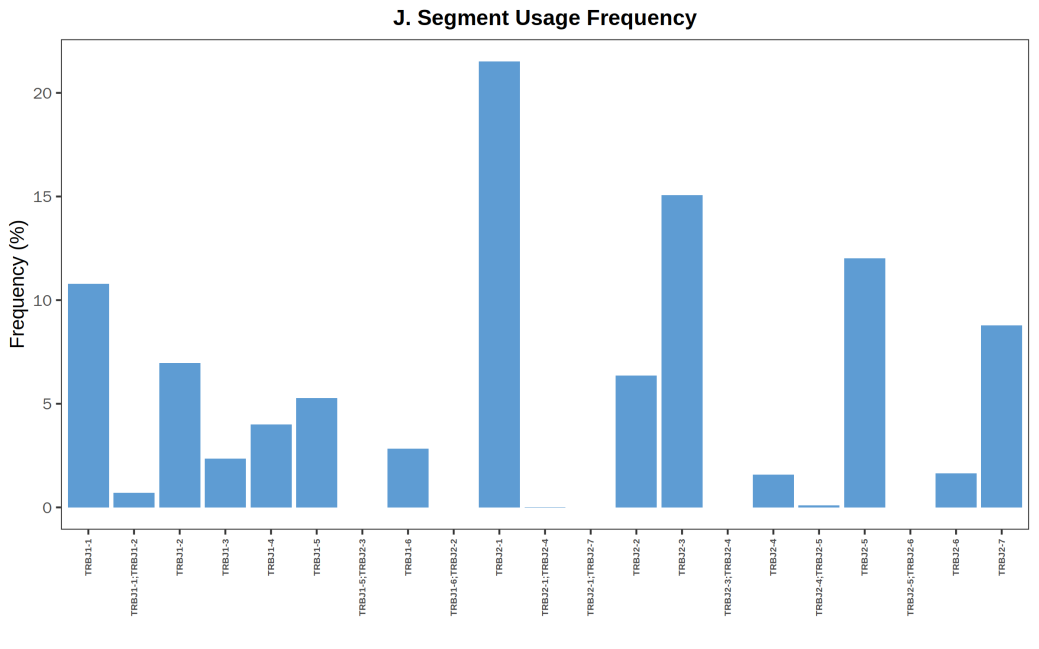
图4 J 基因使用频率分布直方图
上图中横坐标表示不同的 J 片段的名称,纵坐标表示对应 J 片段的使用频率
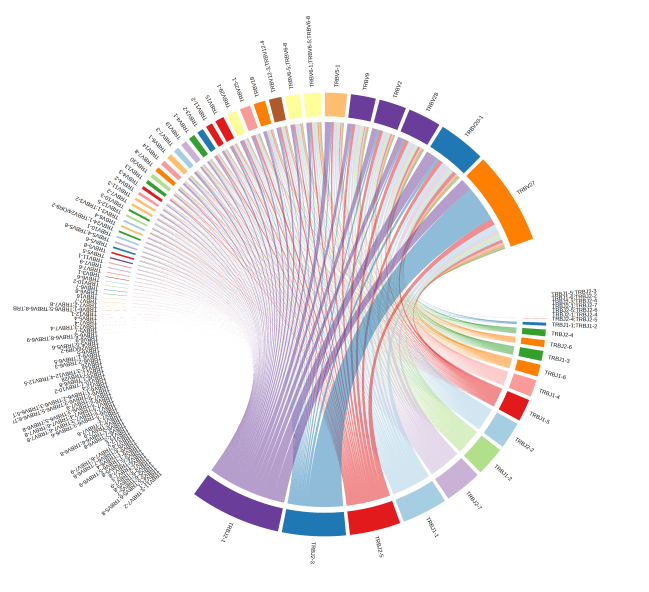
图5 V-J 基因对使用频率分布环形图
上图为 V-J 对使用频率分布环形图。弧对应于不同的 V 片段和 J 片段,按其在样本中的频率进行缩放。丝带代表 V-J 配对,其大小按配对频率缩放
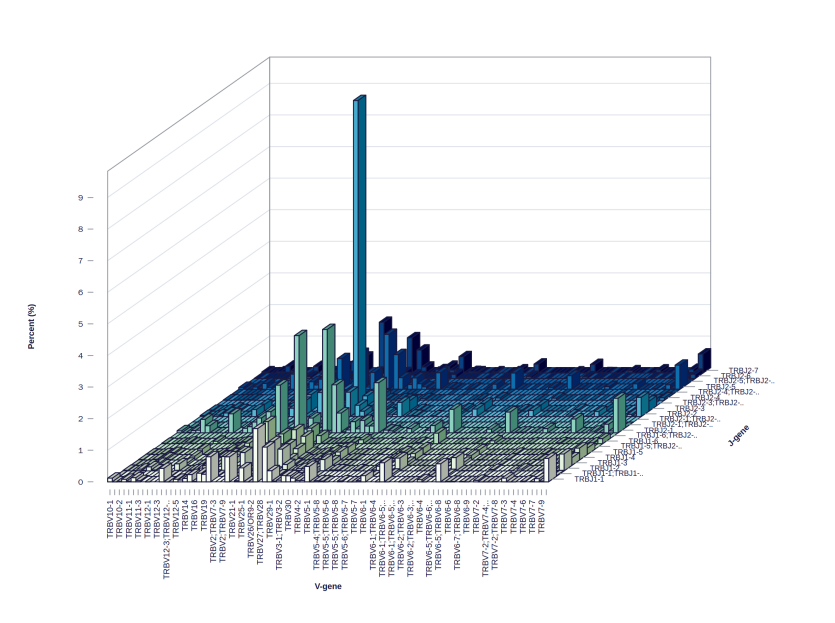
图6 V-J 基因对使用频率分布三维柱状图
上图为 V-J 对三维柱状图。其中 X 轴表示 V 片段的名称,Y 轴表示 J 片段的名称,Z 轴表示克隆频率。
在疾病进程中,TCR V和J基因的使用频率偏好性可能出现显著变化。这是因为疾病状态下,某些特定的T细胞克隆可能因为针对某种抗原的高亲和力而被强烈激活和扩增,从而导致某些V或J基因的使用频率增加。例如,在某些类型的肿瘤中,已发现存在肿瘤特异性T细胞克隆的富集,表现为TCR V和J基因使用频率的非随机性分布。另一方面,在某些疾病相关的抗原刺激下,某些特定的TCR克隆占主导地位时,可能导致TCR多样性下降,使得原本广泛分布的V或J基因使用频率变得集中。
随着年龄的增长,免疫系统经历正常的生理衰老过程,称为免疫衰老。在这个过程中,T细胞库的多样性可能会减少,一部分高频使用的V或J基因组合可能会更频繁地出现,同时罕见的组合可能减少,导致整体TCR多样性降低。
此外,老年个体可能对新抗原的免疫响应减弱,这也可能影响TCR V和J基因的使用频率偏好性,使其倾向于维持以前接触过的抗原的记忆反应,而不是产生新的克隆来应对新挑战。
TCR V和J基因的频率偏好性与疾病进程或年龄的相关性是复杂的,依赖于多种因素,包括个体的遗传背景、环境暴露、疾病类型和严重程度等。
PART 03
小结
在肿瘤过继性免疫治疗中,如CAR-T细胞疗法和TILs免疫疗法,对治疗后的受检者的不同时间节点的样本进行TCR免疫组库测序,统计注射液的TCR克隆残留比例,识别样本间的共有克隆,并追踪不同样本TCR指标的动态变化,可以辅助评估药物的治疗效果。下期文章即将介绍在肿瘤免疫治疗中对时序样本的检测,敬请期待。
熙宁|精翰NGS实验室应用基于多重PCR建库的免疫组库检测方法特异性地扩增TCR/Ig受体链编码基因(TRB、TRD、TRG,以及IgH、IgL、IgK),检测T/B细胞受体基因的克隆性重排,通过专业的生物信息学流程分析识别受体可变区,统计样本的克隆多样性及V/J片段的使用频率,从而揭示个体的免疫表达谱。本检测方法已经经过了完善的性能验证,可以供申办方直接使用。
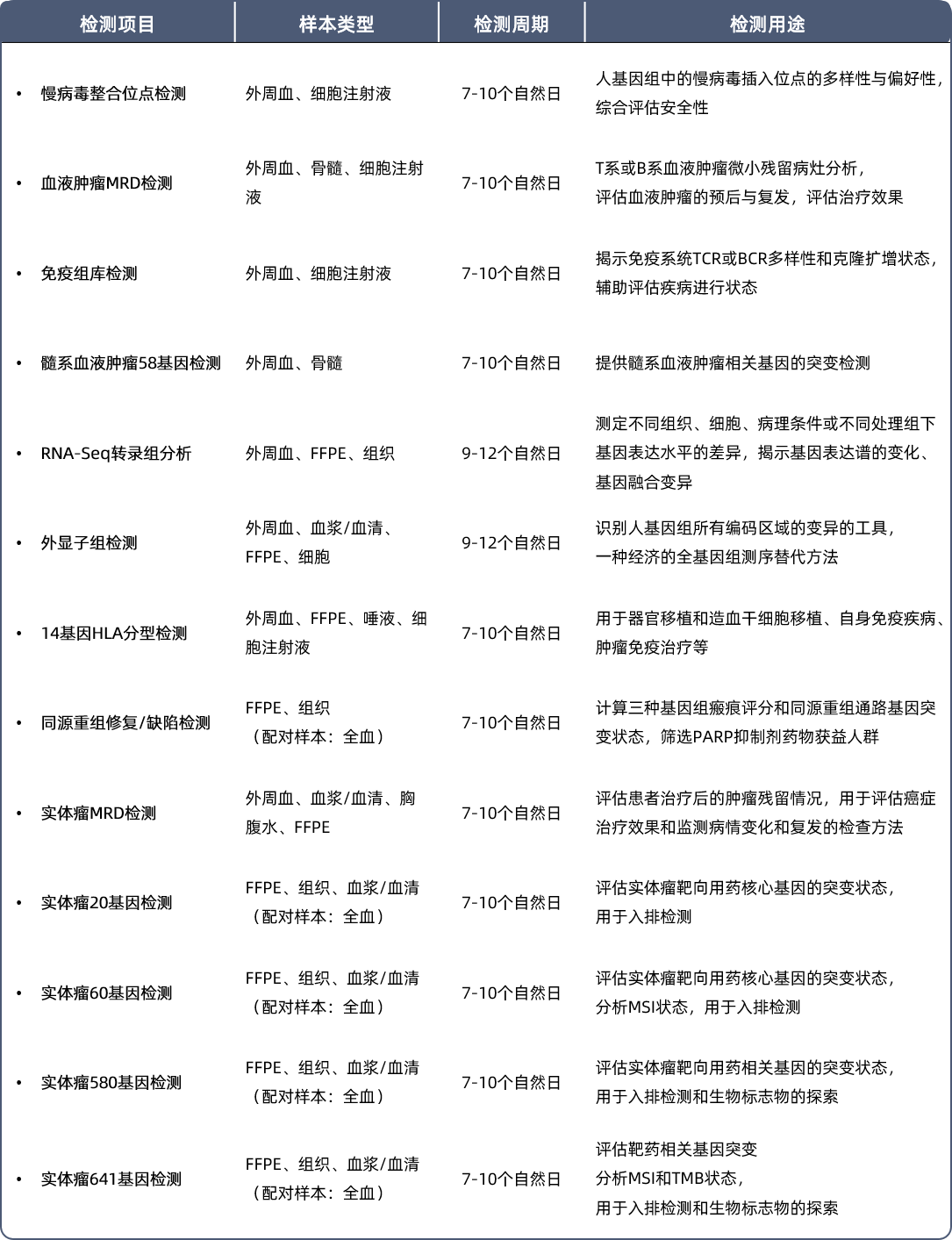
熙宁|精翰NGS实验室依据CAP质量的要求,建立了完善的质量体系。实验室拥有NextSeq CN500测序仪,支持本地测序,且多次满分通过CAP的能力验证。按照质量体系的要求,实验室建立了丰富的检测方法,全面支撑药物的安全性评估、治疗效果评估、入组筛查和生物标志物的探索等,在药物研发的各个环节发挥重要作用:
➢
以慢病毒为载体的CAR-T细胞治疗中,识别有致瘤性的或者其他潜在危险的整合位点,评估整合事件的多样性和偏好性,辅助评估药物潜在的安全性风险;
➢
通过对肿瘤微小残留病灶(MRD)的检测,评估药物治疗效果并提示预后,提前提示复发风险并指导后续干预;
➢
通过基因组和转录组测序,帮助申办方识别与疾病关联的基因突变、融合突变和拷贝数变异等,识别受试者药物靶点变异情况,以及与药物转运、代谢和相关信号通路的基因变化,从而指导患者入排,发现药物研发的潜在靶点和生物标志物,并指导耐药机制研究和新的治疗策略开发;
➢
评估候选药物对基因表达的影响,筛选出对特定靶标有显著作用的化合物,同时通过分析药物处理前后基因表达变化,优化药物的剂量、作用机制和潜在副作用。
欢迎后台留言咨询。
参考文献(上下滑动翻看)
[1] Zhang W, Du Y, Su Z, et al. IMonitor: a robust pipeline for TCR and BCR repertoire analysis[J]. Genetics, 2015, 201(2): 459-472.
[2] Wang T, Wang C, Wu J, et al. The different T-cell receptor repertoires in breast cancer tumors, draining lymph nodes, and adjacent tissues[J]. Cancer immunology research, 2017, 5(2): 148-156.
[3] Chen S Y, Yue T, Lei Q, et al. TCRdb: a comprehensive database for T-cell receptor sequences with powerful search function[J]. Nucleic acids research, 2021, 49(D1): D468-D474.
[4] Shugay M, Bagaev D V, Turchaninova M A, et al. VDJtools: unifying post-analysis of T cell receptor repertoires[J]. PLoS computational biology, 2015, 11(11): e1004503.
[5] Tang J, Pearce L, O'Donnell-Tormey J, et al. Trends in the global immuno-oncology landscape[J]. Nat. Rev. Drug Discov, 2018, 130(176): 112.
[6] Zhao Q, Jiang Y, Xiang S, et al. Engineered TCR-T cell immunotherapy in anticancer precision medicine: pros and cons[J]. Frontiers in immunology, 2021, 12: 658753.
[7] Sarnaik A A, Hamid O, Khushalani N I, et al. Lifileucel, a tumor-infiltrating lymphocyte therapy, in metastatic melanoma[J]. Journal of Clinical Oncology, 2021, 39(24): 2656.
[8] Pai J A, Satpathy A T. High-throughput and single-cell T cell receptor sequencing technologies[J]. Nature methods, 2021, 18(8): 881-892.
[9] Jiang, X., et al., Comprehensive TCR repertoire analysis of CD4(+) T-cell subsets in rheumatoid arthritis. J Autoimmun, 2020. 109: p. 102432.
[10] Liu, X., et al., T cell receptor beta repertoires as novel diagnostic markers for systemic lupus erythematosus and rheumatoid arthritis. Ann Rheum Dis, 2019. 78(8): p. 1070-1078.