



随着科技的不断进步,免疫组库测序方法正为我们提供越来越深入的洞察力,揭示着免疫系统的奥秘。在本文中,我们将深入探讨免疫组库测序的方法和技术,解析其背后的科学原理,以及如何利用这一技术来揭示免疫系统的复杂性。通过了解免疫组库测序的工作流程和应用,我们可以更好地理解人体免疫系统如何应对病原体侵袭、疾病发展以及免疫相关治疗的潜在机制。本文将带领您深入了解免疫组库测序方法的精髓,让我们一起走进这个神奇的科学领域,探寻免疫系统的奥秘之旅!
PART 01
免疫组库检测方法
分析免疫系统的传统方法,如流式细胞术和免疫显微镜光谱分型,存在诸多的局限性,费时费力且费用昂贵[1]。
在下一代测序(NGS)技术之前,最常见的分析免疫受体的方法是Sanger测序,但Sanger测序数据通量较低,成本较高,运行时间较长。与Sanger测序相比,NGS可以以更低的成本、更高的通量和更短的运行时间对免疫系统进行更广泛的检测,因此,NGS技术成为免疫组库检测最常用的技术。NGS免疫组库测序涉及几个过程,包括细胞处理、核酸提取、文库制备、测序和生物信息学分析(图1)。
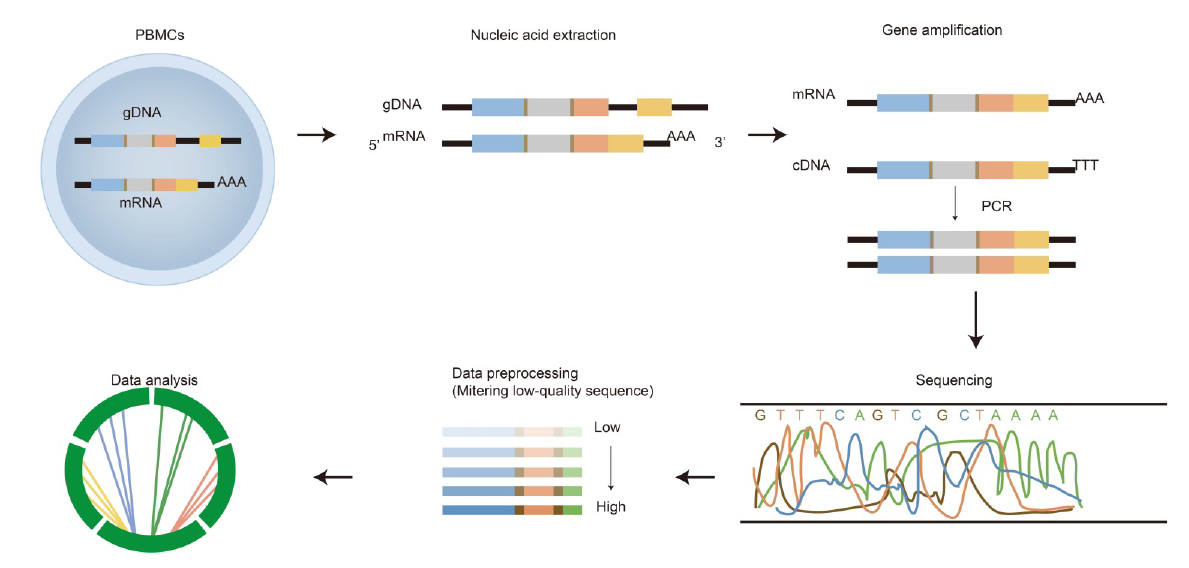
图1 免疫组库测序和分析过程[1]
PART 02
扩增模板选择
文库制备是生成免疫组库测序纯文库的基础,文库制备可以使用gDNA作为模板,使用V和J片段引物,或者使用cDNA作为模板,使用V前导引物、内部V引物、J引物或恒定区引物(图3)。除了体细胞突变对引物结合的潜在影响相关的考虑因素外,两种原料模板各有优劣,主要取决于检测的目标。gDNA与细胞数量成比例相关,因此通常用于计算抗原特异性或靶T/B细胞的比例;而信使核糖核酸与细胞功能/活化密切相关。
gDNA模板的优势是,它允许分析产生蛋白质产物的有效重排基因序列,以及在V(D)J重排过程中非编码重排基因序列,非编码重排基因序列虽然不产生功能蛋白,但提供了Ig基因座重排特征的信息,如基因片段重排频率、核酸外切酶进行的片段消化以及非模板碱基插入水平。这可用于评估与受体基因重排或B细胞选择相关的免疫表型,例如免疫缺陷疾病。每个细胞只有一个有效重排的TCR/BCR基因座,因此gDNA可评估细胞群中T/B细胞克隆的大小。相反,在单个T/B细胞内会产生多个mRNA拷贝,并且TCR/BCR重排基因在不同细胞亚群中mRNA表达水平不同,例如在幼稚细胞中表达较低,在浆母细胞中的高表达,意味着如果只有单个RNA样品可用于分析,则不能可靠地区分具有高TCR/BCR mRNA表达的细胞的存在。然而,使用RNA作为测序起始原料模板还有其他优势,主要是它能够鉴定与特定V(D)J重排相关的重链类型,RNA模板的另一个潜在优势是,它保留细胞样品中存在的T/B细胞克隆的完整性,因为每个B细胞可以贡献其TCR/BCR重排的多个mRNA拷贝,因此,如果在纯化过程中丢失一些模板,样品中代表的T/B细胞克隆的数量不会像gDNA那样线性减少。最后,重排的gDNA片段仅占总DNA量的一小部分,而cDNA每ng扩增的模板含有更多可扩增的cDNA模板。由于可以添加到PCR反应中的DNA模板的量是有限的,因此与gDNA模板相比,使用cDNA模板可以用更少的PCR反应得到更复杂的BCR/TCR重排文库(表1)。
表1 gDNA和mRNA作为文库扩增原料模板的优劣[1]

PART 03
文库构建扩增方法选择
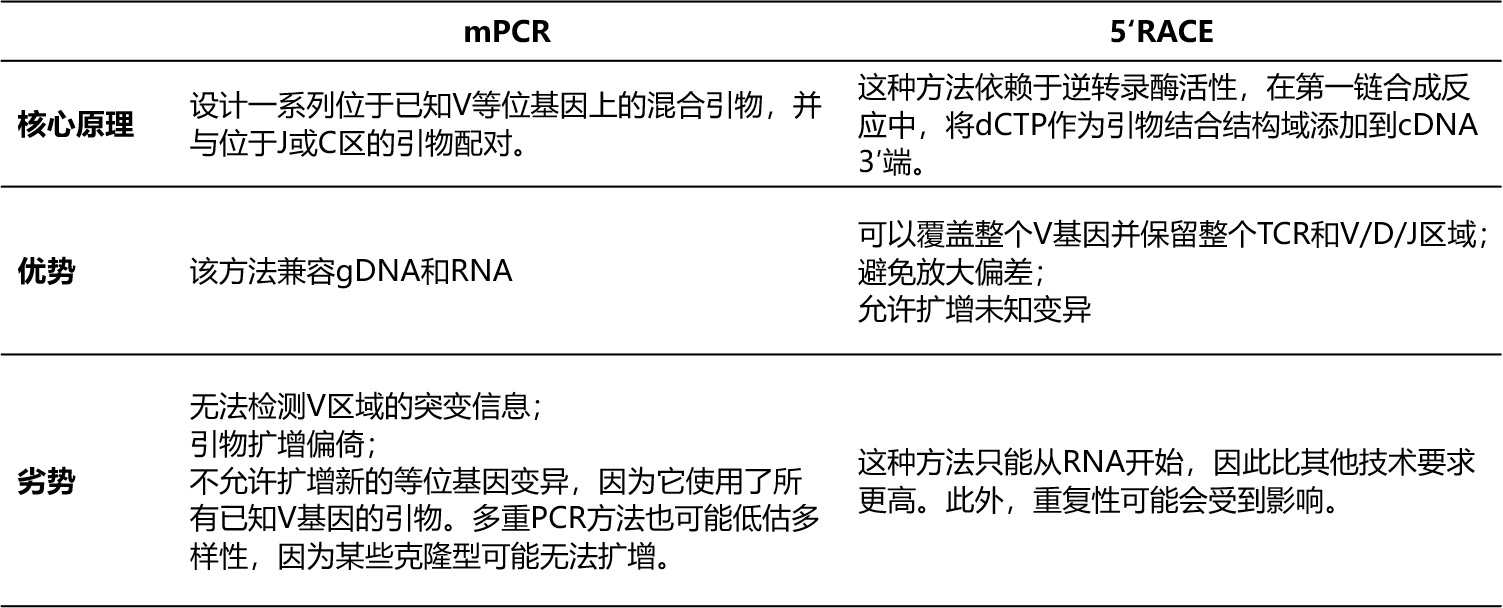
文库制备的扩增方法也是非常重要的,常用的方法有多重聚合酶链式反应(mPCR)和5’RACE。mPCR使用混合引物来捕获多个区域,引物设计主要集中在框架(FR)区域FR1、FR2和FR3上,该区域的突变相对少于CDR区更少,可能是由于框架(FR)区突变破坏抗体结构稳定,导致B细胞在体内的持久性受到影响。这种扩增方法既可用于gDNA,也可用于mRNA。扩增原理与普通聚合酶链式反应相似,只是引物数量增多。但不同引物之间存在效率差异和交叉反应性,导致扩增产物中引入偏差,需要对引物设计和引物浓度进行优化[1]。
5’RACE仅用于捕获mRNA,一组基因特异性引物能够与3’端基因片段互补结合,使用V、J或恒定区内的引物,可以最大限度地减少引物偏倚和多重PCR引入的偏差,同时也有利于扩增引物结合位点发生突变的lg序列。然而,对一个基因座上所有V基因片段分析显示,一些可能具有短的5’非转录区(UTR)(短至30bp),而一些可能具有非常长的5’UTR(高达9kb)(图2,图3)。因此,通过这种方法制作的文库可能偏好于较短的序列,这种方法的扩增建库可能更复杂,需要RNA作为起始材料,缺少基因组DNA重排的分析,检测性能受限于RNA样本总量和质量,以及逆转录酶的效率。总之,这两种方法各有优缺点(表2)。
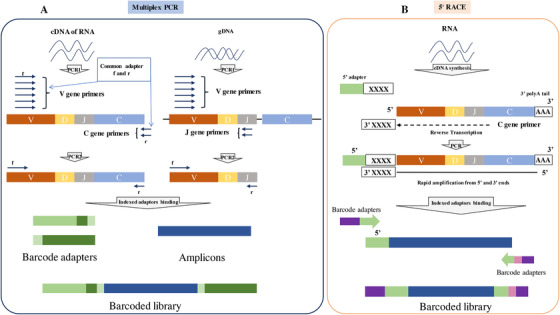
图2 TCR文库构建过程[4]
注:(A)多重聚合酶链式反应的过程
(B) 5‘RACE的过程
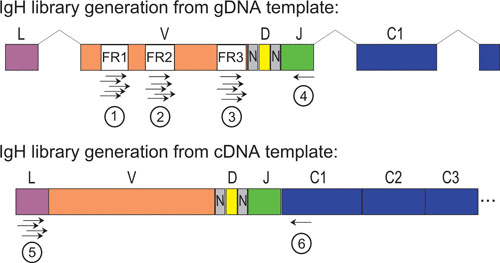
图3 使用gDNA或cDNA生产IgH文库[3]
注:上图显示了编码抗体重链的重排gDNA。左引物设计在框架1、2或3(FR1、FR2、FR3)区域(用带圆圈的数字1、2和3标记),互补右引物设计在与J基因片段互补区域(标记4),PCR可扩增VDJ基因重排。左侧框架引物组显示了多个引物,表明扩增不同家族的V片段所需的不同引物。前导肽外显子通过短内含子与V片段分开。下图显示了由编码重链的成熟mRNA产生的cDNA。与恒定区杂交的引物(标记为6)可以用作5’RACE方案的初始特异性引物,或者可以与前导序列中的引物(标为5)或框架引物一起进行PCR,扩增VDJ基因重排。与VDJ基因重排相关的恒定区不同类型可以在该文库中进行鉴别。
表2 mPCR和5’RACE两种扩增方法的优劣[1]
免疫组库测序是一个复杂的过程,需要专业人员操作以便最大限度地减少测序错误,在文库制备和测序中都可能引入错误,文库制备可能会在核酸提取和PCR扩增过程中引入错误,而测序错误源于不同的测序平台以及测序上机文库的浓度和纯度。为了减少测序错误,可以使用一些校正方法,如聚类算法和独特的DNA条形码技术。聚类算法的是通过将相似的序列分组在一起来降低测序误差,独特的DNA条形码技术是添加独特的分子标签(UMI),调整PCR过程中的误差或扩增偏差[1]。
PART 04
其他免疫组库检测技术
最近的研究中,RNA-seq也用于研究免疫系统,虽然该方法提供了更全面的基因表达信息,但这种方法并不能提供完整的转录组序列,但这对于在更多细胞中鉴定低丰度CDR3序列是必要的,因此,RNA-seq敏感性不足以进行严格的免疫组库分析[1]。另外还有新发展的单细胞测序技术,基于流式细胞术、微流体设备和微孔板来分离单细胞,然后通过具有独特条形码的液滴或具有磁珠的乳液制备文库,进行高通量测序,单细胞分离技术与高通量测序技术相结合,有助于获得单细胞高通量数据,可以剖析B细胞重链:轻链和T细胞α链:β链或δ链:γ链的配对信息,分析特定疾病抗体库,但由于单细胞测序检测成本高、检测流程繁琐、数据解读分析复杂等问题,整体适用范围有限。
本文我们深入介绍了免疫组库测序的方法和技术,解析了其在科学研究领域中的重要性和应用。从测序样本的准备到数据分析的过程,一步步揭示了免疫组库测序技术的精髓和复杂性,展现了这一技术在揭示免疫系统机制中的不可或缺的作用。
下一篇文章将带您进入免疫组库测序在药物研发中的应用领域,探讨免疫组库测序如何在药物研发过程中发挥关键作用,帮助研发人员更好地理解免疫系统在疾病治疗中的作用机制,揭示潜在的药物靶点和治疗策略。
熙宁|精翰NGS实验室应用基于多重PCR建库的免疫组库检测方法特异性地扩增TCR/Ig受体链编码基因(TRB、TRD、TRG,以及IgH、IgL、IgK),检测T/B细胞受体基因的克隆性重排,在时序样本检测中,通过检测患者体内TCR谱系的变化来评估治疗效果以及跟踪疾病进展或复发。本检测方法已经经过了完善的性能验证,可以供申办方直接使用。欢迎您后台留言咨询。
参考文献:
[1] Liu H, Pan W, Tang C, Tang Y, Wu H, Yoshimura A, Deng Y, He N, Li S. The methods and advances of adaptive immune receptors repertoire sequencing. Theranostics. 2021 Aug 19;11(18):8945-8963. doi: 10.7150/thno.61390. PMID: 34522220; PMCID: PMC8419057.
[2] Aran A, Garrigós L, Curigliano G, Cortés J, Martí M. Evaluation of the TCR Repertoire as a Predictive and Prognostic Biomarker in Cancer: Diversity or Clonality? Cancers (Basel). 2022 Mar 31;14(7):1771. doi: 10.3390/cancers14071771. PMID: 35406543; PMCID: PMC8996954.
[3] Boyd SD, Joshi SA. High-Throughput DNA Sequencing Analysis of Antibody Repertoires. Microbiol Spectr. 2014 Oct;2(5). doi: 10.1128/microbiolspec.AID-0017-2014. PMID: 26104353.
[4] Li N, Yuan J, Tian W, Meng L, Liu Y. T-cell receptor repertoire analysis for the diagnosis and treatment of solid tumor: A methodology and clinical applications. Cancer Commun (Lond). 2020 Oct;40(10):473-483. doi: 10.1002/cac2.12074. Epub 2020 Jul 17. PMID: 32677768; PMCID: PMC7571402.
[5] Joshi K, Milighetti M, Chain BM. Application of T cell receptor (TCR) repertoire analysis for the advancement of cancer immunotherapy. Curr Opin Immunol. 2022 Feb;74:1-8. doi: 10.1016/j.coi.2021.07.006. Epub 2021 Aug 25. PMID: 34454284.
[6] 12 Zhang S, Yang T, Liu X, Yang J, Zheng X. Antibody repertoire sequencing analysis. Acta Biochim Biophys Sin (Shanghai). 2022 May 25;54(6):864-873. doi: 10.3724/abbs.2022062. PMID: 35713313; PMCID: PMC9828323.
[7] Nixon AB, Schalper KA, Jacobs I, Potluri S, Wang IM, Fleener C. Peripheral immune-based biomarkers in cancer immunotherapy: can we realize their predictive potential? J Immunother Cancer. 2019 Nov 27;7(1):325. doi: 10.1186/s40425-019-0799-2. PMID: 31775882; PMCID: PMC6880594.
[8] Schrama D, Ritter C, Becker JC. T cell receptor repertoire usage in cancer as a surrogate marker for immune responses. Semin Immunopathol. 2017 Apr;39(3):255-268. doi: 10.1007/s00281-016-0614-9. Epub 2017 Jan 10. PMID: 28074285.
[9] Porciello N, Franzese O, D'Ambrosio L, Palermo B, Nisticò P. T-cell repertoire diversity: friend or foe for protective antitumor response? J Exp Clin Cancer Res. 2022 Dec 22;41(1):356. doi: 10.1186/s13046-022-02566-0. PMID: 36550555; PMCID: PMC9773533.