



一、背景介绍
DNA和组蛋白通过调节亲和性和功能来进行修饰,这些修饰的改变是肿瘤的标志之一。表观遗传治疗致力于促进或维持恶性表型的DNA甲基化模式或组蛋白翻译后修饰正常化。近年来,组蛋白的乙酰化修饰是表观遗传学领域的热门研究方向。研究者发现,组蛋白乙酰化是与机体生理功能及病理表征联系最为紧密的修饰化之一。HDACs和HATs 等去乙酰化或乙酰化酶靶点是表观遗传抗肿瘤领域研究的热门靶点,在相关药物的临床试验中,组蛋白乙酰化水平的变化是药物疗效的一个重要指标。
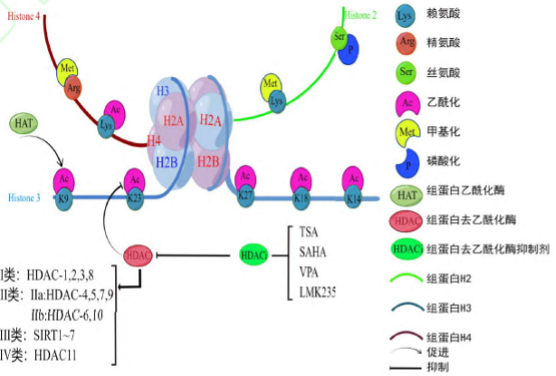
图1. 组蛋白乙酰化及去乙酰化酶修饰相关酶分子简介
二、流式检测组蛋白乙酰化的基本原理
一般认为,组蛋白乙酰化修饰多数发生在组蛋白H3或H4N端特定的赖氨酸残基上,目前市面上已有很多抗乙酰化组蛋白H3和H4直接标记的流式抗体。临床相关的组蛋白乙酰化Marker有很多,如H3K9ac、H3K14ac和H4K5ac等,使用对应Marker的抗体进行标记,通过流式检测某荧光素对应通道的信号值或信号比值(如H3K9ac/H3),来反映细胞组蛋白乙酰化的强弱。
三、关于模拟临床样品的设计考量
1.生物基质的选择
考虑到临床分析,该检测需采用全血或分离的PBMC进行。
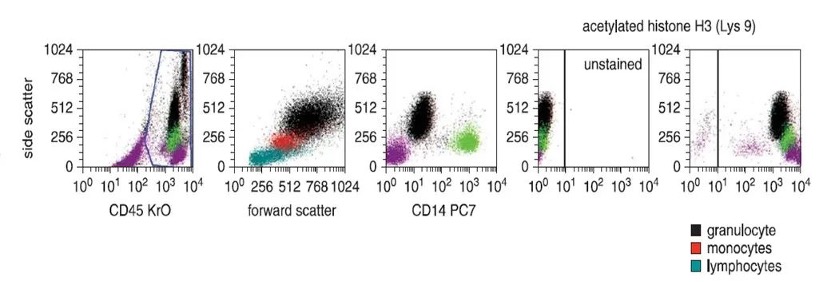
图2. 全血样本白细胞中H3K9乙酰化水平
2.不同水平组蛋白乙酰化模拟临床样品的制备
在全血或PBMC体外加药处理调节组蛋白乙酰化表达水平,生成高表达或低表达的模拟临床样品,处理不同时长后,获取各个时间点的样品进行流式细胞术检测。
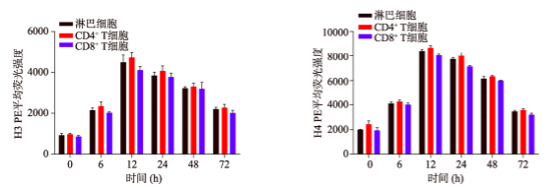
图3. 西达本胺(HDACI)处理不同时长,
PBMC组蛋白乙酰化水平的变化
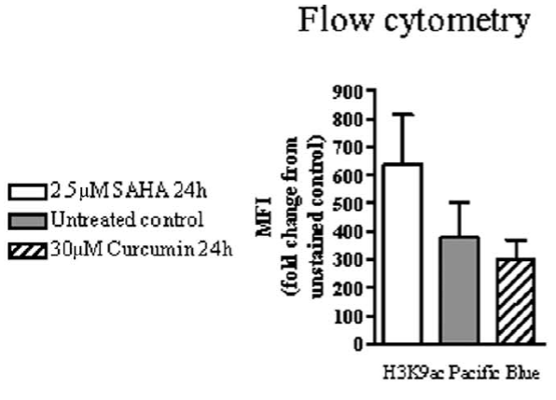
图4. 不同药物(包含HDACI和HATI)
处理后H3K9ac水平的变化
四、关于方案设计要点的考量
1.储存条件对组蛋白乙酰化水平的影响
据文献报道,全血样品可在临床中心采集后尽量24h(建议不超过48h)内于4°C运输至检测中心,尽快裂红或裂红固定,裂红后的细胞尽快-80°C保存,裂红固定后的细胞保存不超过一周,进行流式检测分析,文献结果如图5。
该检测可考虑熙宁生物自主产权的冻存全血的方法,无需裂红,可直接冻存。
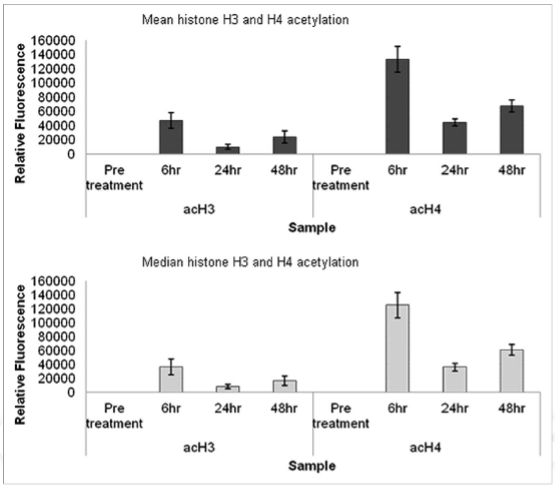
图5. 患者给药LBH589(HDACI)后,采集全血,裂红固
定,流式细胞术检测体内白细胞组蛋白乙酰化水平的变化
2.固定、破膜或破膜固定剂的选择对染色效果的影响
由于不同的固定、破膜或破膜固定剂的成分可能会影响抗体的结合以及导致荧光素分子降解,从而影响信号强度,甚至检测不到阳性信号,因此需要选择合适的固定、破膜或破膜固定剂。
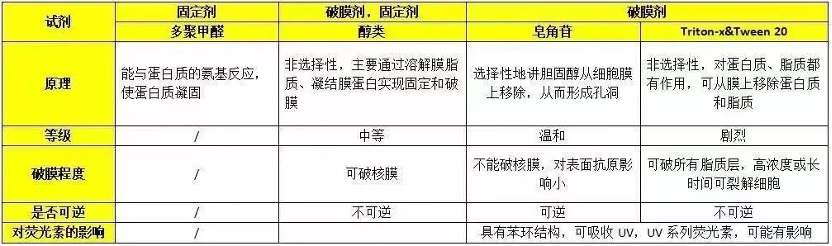
图6. 固定破膜试剂基本信息汇总
五、熙宁优势
蛋白的磷酸化、甲基化、乙酰化、ADP-核糖化修饰和泛素化等共价修饰的变化是细胞生理状态,机体健康状态,药物是否发挥药效的重要指标。在药物临床研究阶段,检测蛋白翻译后修饰水平,评估给药前后的变化情况,成为临床阶段一些小分子药物的主要药效学(PD)指标之一,可用于指导创新药物二期临床试验的用药剂量。
熙宁生物流式细胞转化医学团队,具有10余年药物开发和生物标志物检测经验,并成功开发了超100种不同的生物标志物方法,用于临床阶段药物安全性和有效性的评估,其中包括一系列基于流式平台、ELISA和MSD平台的磷酸化蛋白、甲基化蛋白、ADP-核糖化修饰、泛素化和乙酰化蛋白的检测方法,助力药物临床研究。欢迎各位新老朋友,前来探讨您感兴趣的靶点和检测方法。
参考文献
1.Bates, Susan E. "Epigenetic therapies for cancer." New England Journal of Medicine 383.7 (2020): 650-663.
2.Watson, Maria, et al. "The study of epigenetic mechanisms based on the analysis of histone modification patterns by flow cytoametry." Cytometry Part A 85.1 (2014): 78-87.
3.韩韦钰, et al. "组蛋白去乙酰化修饰调控心血管疾病的发生与发展." 中国组织工程研究 27 (2023): 1-7.
4.Watson, Maria, and David Hedley. "Whole blood measurement of histone modifications linked to the epigenetic regulation of gene expression." Current Protocols in Cytometry 71.1 (2015): 6-36.
5.李建辉, et al. "流式细胞术检测组蛋白乙酰化水平方法的建立与应用." 遗传 38.6 (2016): 581-587.
6.Glaser, Keith B., et al. "Gene expression profiling of multiple histone deacetylase (HDAC) inhibitors: defining a common gene set produced by HDAC inhibition in T24 and MDA carcinoma cell lines." Molecular cancer therapeutics 2.2 (2003): 151-163.
7.Rigby, Lin, et al. "Methods for the analysis of histone H3 and H4 acetylation in blood." Epigenetics 7.8 (2012): 875-882.
8.Obier, Nadine, and Albrecht M. Müller. "Chromatin flow cytometry identifies changes in epigenetic cell states." Cells Tissues Organs 191.3 (2010): 167-174.